빅분기 실기 최종 정리 3 - 통계 검정
https://wikidocs.net/287831
검정
귀무가설 : “변화 X”, “차이 X”, “관계 X” ,"영향없다","같다","우연"
H0 : 귀무
p<0.05 기각 : 먼가이슈가잇는거임, 차이가크거나 관계가잇거나
from scipy.stats import shapiro, levene, ttest_ind,chi2_contingency,chisquare,pearsonr
z검정(단일표본)
https://wikidocs.net/287750
from scipy.stats import norm
import numpy as np
# <span id="주어진-값"></span>주어진 값
x_bar = 5.1 # 표본 평균
mu_0 = 5.0 # 귀무가설 하 모집단 평균
sigma = 0.4 # 모집단 표준편차
n = 40 # 표본 수
# <span id="z-통계량-계산"></span>z-통계량 계산
z = (x_bar - mu_0) / (sigma / np.sqrt(n))
# <span id="양측-검정이므로-p-value는-양쪽-꼬리-합"></span>양측 검정이므로 p-value는 양쪽 꼬리 합
p = 2 * norm.sf(abs(z)) # 또는 p = 2 * (1 - norm.cdf(abs(z)))
print(f'z 통계량: {z:.3f}')
print(f'p-value: {p:.3f}')
t검정
| 종류 |
비교 대상 |
예시 |
Python 함수 |
자유도 |
| 독립 표본 t-검정 |
서로 다른 집단 |
남 vs 여 평균 키 비교 |
ttest_ind(g1,g2) |
n-1 |
| 대응 표본 t-검정 |
같은 사람/같은 객체 전후 비교 |
다이어트 전 vs 후 |
ttest_rel(befor ,after) |
n1 + n2 − 2 |
| 단일 표본 t-검정 |
기준값(하나의 평균)과 비교 |
우리반 키가 전국 평균과 같나? |
ttest_1samp(g,기준) |
n-1 |
흐름
import pandas as pd
from scipy.stats import shapiro, levene, ttest_ind
# <span id="예시-컬럼-이름과-그룹-값"></span>예시: 컬럼 이름과 그룹 값
group_col = 'group' # 예: 성별, 반, 처리군 등
value_col = 'value' # 예: 점수, 키, 혈압 등
g1_name = 'A'
g2_name = 'B'
# <span id="1-데이터-나누기"></span>1) 데이터 나누기
group1 = df[df[group_col] == g1_name][value_col]
group2 = df[df[group_col] == g2_name][value_col]
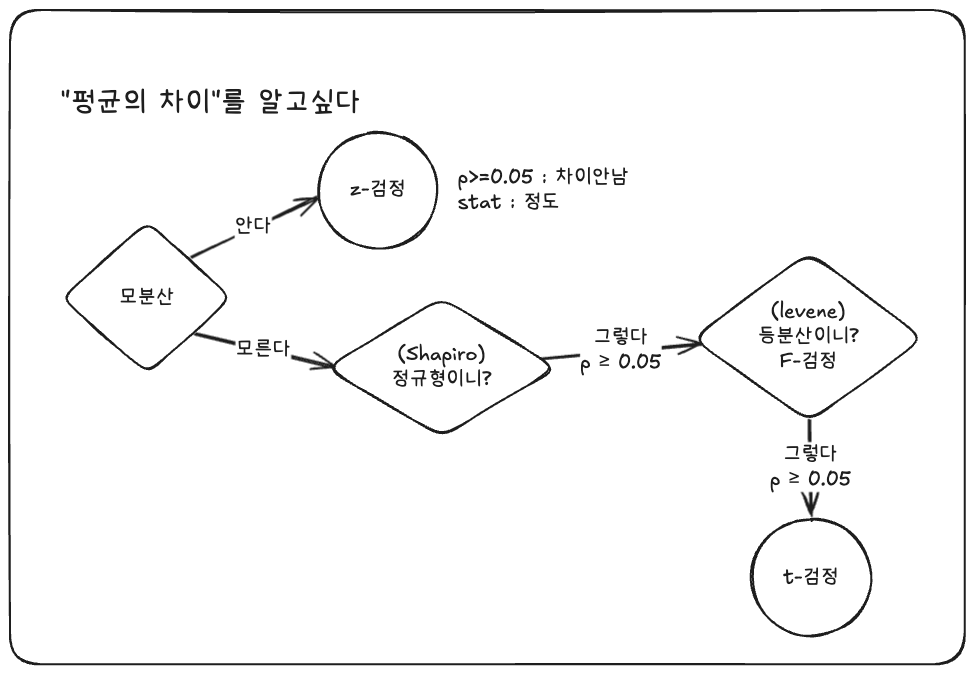
print("=== 1. 정규성 검정 (Shapiro-Wilk) ===")
stat1, p1 = shapiro(group1) # stat1 클수록 정규형에 가까움
stat2, p2 = shapiro(group2)
print(f"{g1_name} Shapiro p-value:", p1)
print(f"{g2_name} Shapiro p-value:", p2)
if p1 >= 0.05 and p2 >= 0.05:
print("→ 두 집단 모두 정규성 가정 만족 (p ≥ 0.05)")
else:
print("→ 정규성 가정 일부/전체 불만족 (p < 0.05) - 해석 시 주의")
print("\n=== 2. 등분산 검정 (Levene) ===")
stat_levene, p_levene = levene(group1, group2)
print("Levene F-statistic:", stat_levene)
print("Levene p-value:", p_levene)
if p_levene >= 0.05:
print("→ 등분산 가정 만족: equal_var=True 사용")
equal_var_flag = True
else:
print("→ 등분산 가정 불만족: equal_var=False 사용 (Welch t-test)")
equal_var_flag = False
print("\n=== 3. 독립 표본 t-검정 (ttest_ind) ===")
t_stat, p_t = ttest_ind(group1, group2, equal_var=equal_var_flag)
print("t-statistic:", t_stat)
print("t-test p-value:", p_t)
alpha = 0.05
if p_t < alpha:
print(f"→ 유의수준 {alpha}에서 두 집단의 평균은 다르다 (귀무가설 기각).")
else:
print(f"→ 유의수준 {alpha}에서 두 집단의 평균은 같다 (귀무가설 채택).")
카이제곱 검정
독립성 검정
import pandas as pd
from scipy.stats import chi2_contingency
# <span id="교차표빈도표-생성"></span>교차표(빈도표) 생성
table = pd.crosstab(df['성별'], df['브랜드'])
# <span id="카이제곱-독립성-검정"></span>카이제곱 독립성 검정
chi2, p, dof, expected = chi2_contingency(table)
print("Chi2:", chi2) # 관찰값과 기대값이 얼마나 다른가,
print("p-value:", p)
print("dof:", dof)
print("Expected:\n", expected)
# <span id="해석"></span>해석
if p < 0.05:
print("유의수준 0.05에서 두 변수는 관계있") # 귀무가설 기각
else:
print("유의수준 0.05에서 두 변수는 관계가없다(독립).") #귀무가설 채택
| 이름 |
의미 |
키워드 |
|
chi2 |
관찰-기대한 차이 크기 |
차이정도 |
chi2큼 : 독립아님 |
p |
그 차이가 우연인지 확률 |
유의성 |
p < 0.05 : 독립아님 |
dof |
카이제곱 분포 기준선(자유도) |
구조 |
(행−1) × (열−1) |
expected |
기대되는 표 |
비교기준 |
|
적합도 검정
import numpy as np
from scipy.stats import chisquare
# <span id="관측값"></span>관측값
observed = np.array([60, 20, 20])
# <span id="기대비율을-실제-기대빈도로-변환"></span>기대비율을 실제 기대빈도로 변환
expected_ratio = np.array([0.5, 0.3, 0.2])
expected = expected_ratio * observed.sum()
chi2, p = chisquare(f_obs=observed, f_exp=expected)
print("Chi2:", chi2)
print("p-value:", p)
if p < 0.05:
print("유의수준 0.05에서 예상대로안나옴(적합하지 않다).")
else:
print("유의수준 0.05에서 예상대로나옴(적합하다).")
피어슨 검정
선형관계 검정
from scipy.stats import pearsonr
x = df['height']
y = df['weight']
r, p = pearsonr(x, y)
print("r:", r)
print("p-value:", p)
if p < 0.05:
print("→ 유의한 상관관계가 있다.")
else:
print("→ 유의한 상관관계가 없다.")
회귀분석
| 종속변수 |
모델 |
함수 |
| 연속형 |
다중선형회귀 |
sm.OLS |
| 범주형 |
로지스틱회귀(이진/다중) |
sm.Logit, MNLogit |
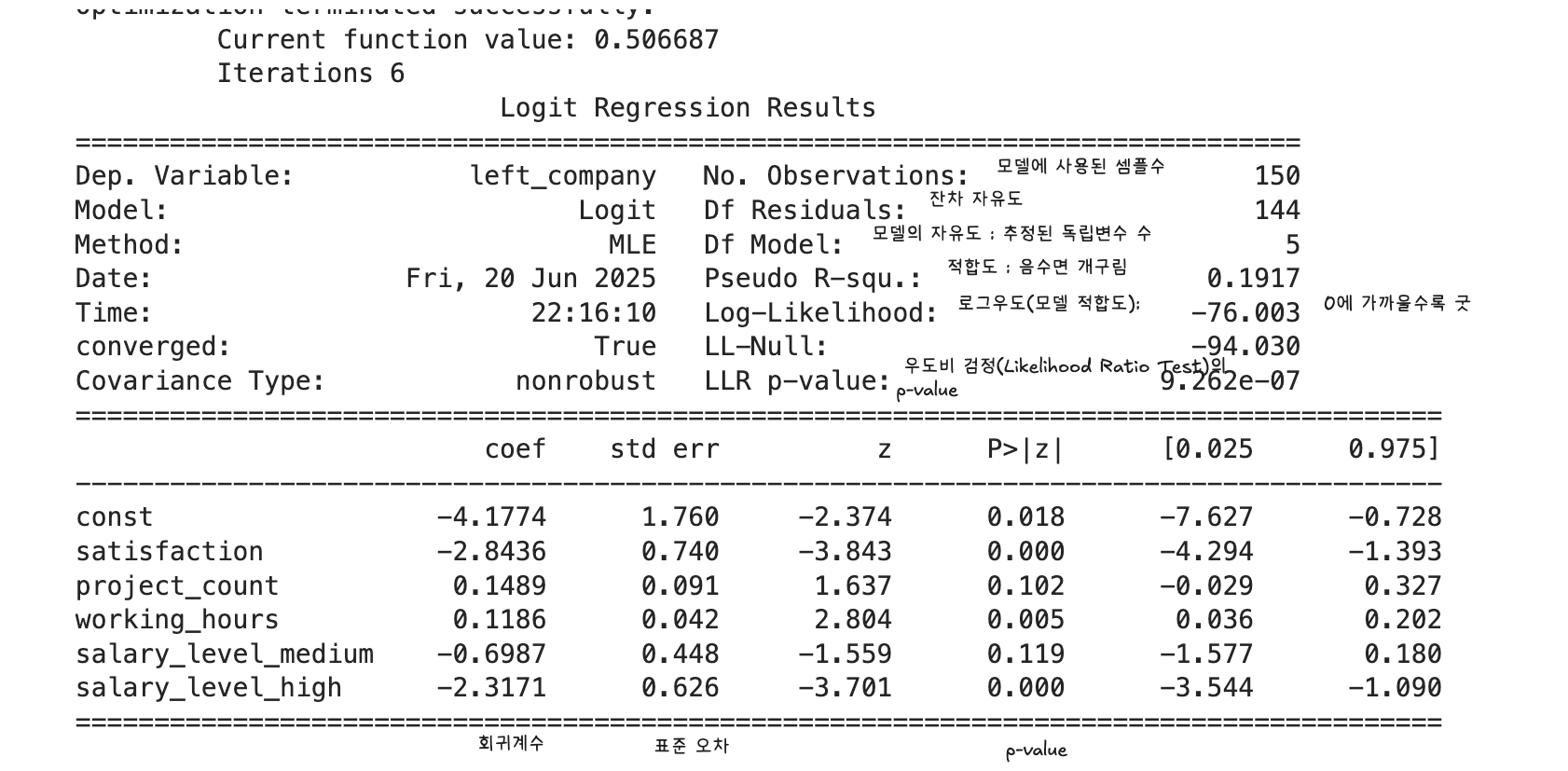
import statsmodels.api as sm
import pandas as pd
X = df.drop(columns=['churn'])
y = df['churn']
X_const = sm.add_constant(X)
model = sm.Logit(y, X_const).fit()
df = pd.DataFrame({
'X1': [1, 2, 3, 4, 5],
'X2': [2, 3, 2, 5, 4],
'Y': [3, 4, 6, 7, 8]
})
X = df<a href="/pages/'X1'%2C-'X2'.html" class="wiki-link">'X1', 'X2'</a> # 독립변수
X_const = sm.add_constant(X) # 절편 추가
y = df['Y'] # 종속변수
model = sm.OLS(y, X_const).fit()
print(model.summary())
- odds_ratios = np.exp(model.params["속성"])
- coef = model.params["속성"]